与蛋白质相比,RNA结构总体来讲是不稳定的,但是我们也可以找到一些相对稳定的结构,对这些结构进行干扰会显著影响下游的生命活动。从这个角度来讲,RNA结构完全可能成为药物开发的靶点,只不过会面临更多的挑战。
10月14日,第5期AIR学术工作坊第三位报告嘉宾:清华大学生命科学学院、清华-北大生命科学联合中心研究院员、博导张强锋教授,为我们做了题为《利用深度神经网络预测小分子与RNA靶标的相互作用》(SmrtNet: Predicting small molecule and RNA target interactions using deep neural network)的报告。
张强锋,2000年于中国科学技术大学获得学士学位;
2006年于中国科学技术大学获得计算机科学与技术博士学位;
2012年于哥伦
比亚大学获得生物化学和分子生物物理博士学位;
2012-2015年于斯坦福大学医学院从事博士后研究工作;
2015年加入清华大学生命科学学院。
现任清华大学生命科学学院、清华-北大生命科学联合中心研究院员、博导,获得国家杰出青年科学基金,中国生化协会2022年第五届普洛麦格生物化学奖。
主要从事结构生物学、基因组学、人工智能和大数据交叉领域研究。
以通讯作者身份在Cell等国际学术刊物发表SCI论文40余篇,工作入选2019、2021年中国生物信息学十大进展,获得赛诺菲-Cell Research 2021年度杰出论文奖。
现任中国生物信息学学会(筹)人工智能与生命科学专委会主任。
张强锋教授在讲座中介绍了一个新兴的研究方向——针对RNA靶标的小分子药物设计,并介绍了其课题组在RNA靶标与小分子的相互作用预测上所取得的最新进展。
传统的药物研发过程主要针对蛋白质靶标进行药物设计,但目前靶标耗尽等问题导致针对蛋白质的药物研发竞争激烈且成功率低。如果将目光投向蛋白质的上游——RNA,并在RNA的阶段对靶标进行调控,也许可以起到同样的效果,并解决蛋白质不可成药的问题。但在此前很长一段时间内以RNA作为药物靶标并未受到广泛的关注,这是由于大家普遍认为RNA在细胞中具有动态、结构不稳定等缺点,而药物的靶标必须具有稳定且特异性的结构。张强锋教授团队长期深耕RNA结构研究,认为RNA中仍然可以找到一些相对稳定,且合适作为药物靶标的结构。此外,Protein Data Bank数据库中,已存在一些小分子与RNA结合的数据,这也为研究RNA-小分子的相互作用提供了重要参考。
张强锋教授进一步举例说明了RNA-小分子结合对生命过程调控的机理。例如,肌萎缩性侧索硬化1型(Myotonic Dystrophy Type 1, DM1)是一种由于DMPK 基因中CTG重复引起的遗传性疾病。这种扩增导致RNA产物形成复杂的发夹结构,并招募MBNL1干扰其发挥正常的剪切和调控功能。研究人员利用小分子直接靶向重复扩增的RNA,将MBNL1从RNA重复区释放出来,从而恢复其功能。如果小分子可以和RNA结构进行结合,尤其是结合RNA与蛋白质复合物的界面,则能够在下游生物过程中发挥重要的调控作用。基于以上分析,张强锋教授指出RNA可以作为小分子药物靶标的机遇和挑战。针对RNA与蛋白质的不同之处,我们需要在深入了解RNA的基础上开发新的药物设计方法。
实际上,在过去的20年中靶向RNA的小分子药物已经初露头角,但目前主流的策略还是利用反义寡核苷酸(ASO),小干扰RNA(siRNA)等与RNA靶标进行结合,从而治疗相关疾病。与其它大分子药物相比,小分子药物具有稳定性好、透膜性强、可以口服、免疫原性弱等优点,是一个极具应用前景的药物研发方向。张强锋教授介绍了目前市场上首个、也是唯一一个被FDA批准的靶向RNA的小分子药物——Risdiplam。Risdiplam被用于治疗神经肌肉萎缩症,其患者的SMN1基因的表达过程存在异常。Risdiplam的药物作用机理是干预SMN2基因对应的RNA剪切过程,促使SMN2基因保留exon7进而产生更多的全长、功能性的SMN蛋白,以弥补SMN1基因的功能损失。除了Risdiplam以外,目前还有三个药物正在临床试验阶段。
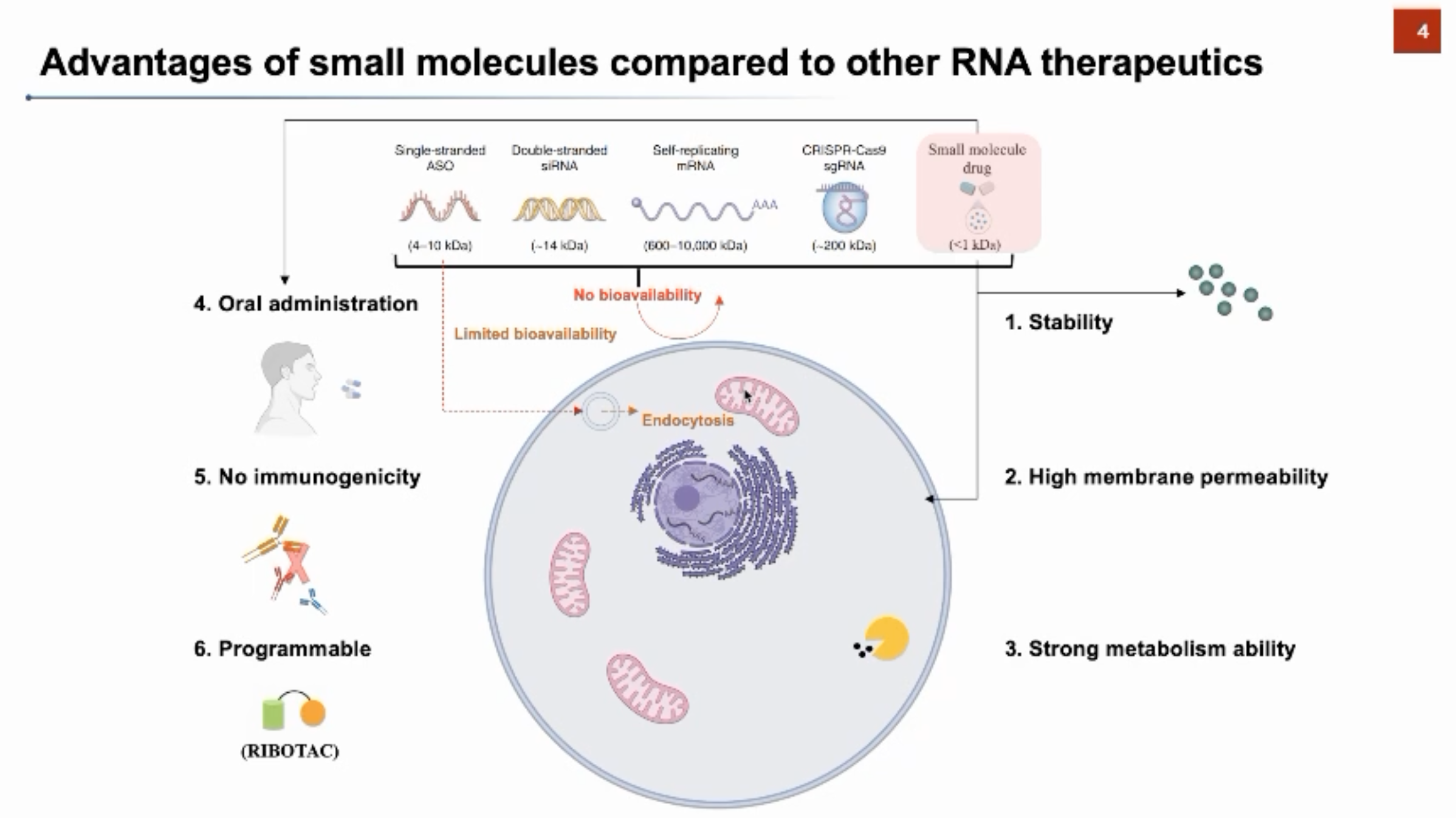
RNA药物设计的核心问题是针对RNA结构靶标找到有特异性相互作用的小分子。目前在湿实验上进行RNA-小分子的相互作用探测的方法(如MST、ALIS、2DCS等)面临实验通量较小、费时费力、且价格昂贵等问题。因此,AI技术对于驱动高通量RNA靶向药物筛选有着广阔的前景。张强锋教授首先回顾了已有针对靶向RNA的小分子的机器学习算法。前人的算法可以总结为三类:1)预测某一个RNA上与小分子结合的靶点,2)预测哪一类小分子可以与核酸(RNA与DNA)进行结合,3)预测与给定RNA三级结构结合的小分子的分子指纹。与前人的工作不同,张强锋教授近期领导团队开发的SmrtNet可以利用深度神经网络高通量地预测小分子与RNA靶点的相互作用,并对小分子与RNA的结合界面提供可解释性分析。
张强锋教授从模型架构、计算表现、文献分析和湿实验验证几个方面介绍了SmrtNet。在模型架构上,SmrtNet对小分子和RNA的序列和结构信息分别进行了建模:在序列方面,分别使用ncRNA-LM和MolFormer两个语言模型对RNA和小分子的序列进行建模;在结构方面,SmrtNet还对RNA的二级结构和小分子的分子图结构分别利用不同的神经网络进行编码。同时,团队还提出了一种新颖的特种融合方法,进一步增强了模型对于小分子和RNA互作信息的学习。团队从Protein Data Bank中提取了1300多个RNA结构,并根据小分子的种类进行训练/验证/测试集的切分。在预测准确率上,SmrtNet可以达到0.81的AUROC。团队还进行了严谨的消融实验和模型架构分析,证明了序列和结构信息都对模型表现起到了重要贡献。在与同类计算方法的比较中,SmrtNet可以取得显著的提升。
团队还对已发表的研究进行了系统性调研,收集了文献中报道过且经过实验验证的132个RNA-小分子相互作用对,形成了一个在模型训练中完全没有出现过的测试数据集。对于该数据集,团队使用SmrtNet进行了相互作用预测,取得了0.89的准确率、0.95的精准率、0.88的召回率、0.89的F1-score、0.88的AUROC、0.94的AUPRC。同样在该数据集上的消融实验表明SmrtNet中的序列信息、结构信息、以及特征融合模块都为最终效果提供了至关重要的贡献。
团队接下来利用梯度反传的方法分析RNA中哪些位点对RNA-小分子的结合有较大的贡献,以此判断小分子在RNA上的结合位点,并在一些典型案例上验证了模型可解释性的合理性。例如Disney研究团队发现了可以结合MYC-IRES的小分子,下图中SmrtNet利用文献中的小分子和RNA,通过模型可解释性预测出的结合位点和文献中的报道高度吻合。在CUG_repeat、HCV_IIa、SNCA-IRE、pre-miR21等RNA靶标上,SmrtNet对结合位点预测均得到了文献中实验结果的支撑,表明该模型可以实现单核苷酸分辨率的结合位点预测。
张强锋教授最后介绍了SmrtNet针对五个RNA靶标的湿实验验证结果。团队针对两个小分子库使用SmrtNet进行虚拟筛选,并对每个靶标都找到了一些可能结合的小分子。根据相关体外实验验证,所筛选出的超过40%的小分子都可以与靶标有效结合,证明了SmrtNet在预测RNA-小分子相互作用上的可靠性。特别地,针对与癌症相关的非常重要的基因MYC,团队找到了一些前人未报道过的可以与MYC-IRES结合的小分子,并通过模型可解释性获得了小分子在该RNA上的结合位点,同时进行了相关的体外实验验证,进一步证明了SmrtNet在预测结合位点上的可靠性。张强锋教授透露与该MYC-IRES相关的更多细胞实验和脱靶效应实验仍在推进之中。期待SmrtNet可以在针对RNA靶标的小分子药物研发中发挥重要的作用。


