蛋白质在生物体中的地位举足轻重,发挥着不可替代的功能。蛋白质的结构和功能联系紧密,如何预测蛋白质的结构?这是我们想探索的终极问题。
——许锦波
活动概况
6月10日上午,第五期AIR学术沙龙在清华大学智能产业研究院(AIR)图灵报告厅如期展开。本期活动荣幸地邀请到了美国芝加哥丰田计算技术研究所教授许锦波教授为我们线上做题为《基于深度学习的蛋白质结构预测》的报告。

主持人马维英介绍许锦波教授
本次讲座由清华大学智能产业研究院(AIR)惠妍讲席教授、首席科学家马维英博士主持,现场观众认真听取报告并与许锦波教授进行了热烈的研讨与思想碰撞,贡献了一场火花四射的思想盛宴。

张亚勤院长向许锦波教授提问
同时,本次讲座也在清华大学智能产业研究院(AIR)微信视频号、哔哩哔哩双平台同步直播。本次活动汇聚了来自清华大学、南洋理工大学、北京大学、中科院、北京航空航天大学、北京协和医学院、华东理工大学,以及智源人工智能研究院、百度研究院、钟鼎资本、NVIDIA、字节跳动等学校及企业单位的专家学者、算法工程师等的参与,全网观看直播人数 2100人次。
讲者介绍

许锦波博士现任美国芝加哥丰田计算技术研究所教授,《Bioinformatics》以及《Journal of Computational Biology》编委;毕业于中国科学技术大学计算机系,中国科学院计算技术研究所和加拿大滑铁卢大学,并在美国麻省理工学院数学系和人工智能实验室接受博士后训练。许博士的研究兴趣包括人工智能和机器学习、优化算法和计算生物学。他于 2016 年首次证明了深度学习可以大大提高蛋白质结构预测的性能,引领了人工智能在蛋白质 结构预测领域的突破。许博士获得了美国斯隆研究奖,美国自然科学基金早期职业奖,《PLoS Computational Biology》创新突破奖,国际计算生物学顶级会议 RECOMB 最佳论文奖和时间检验 奖 (Test-of-Time Award);并多次接受英美杂志如《纽约时报》《经济学人》《财富》《自然》 和《科学》的采访。许博士也是2021年未来科学大奖的主讲嘉宾和2022年在瑞典的诺贝尔论坛特邀嘉宾。
报告内容
蛋白质在生物体中的地位举足轻重,发挥着不可替代的功能。蛋白质的结构和功能联系紧密,如何预测蛋白质的结构?这是我们想探索的终极问题。几十年来,蛋白质的结构预测领域从传统的物理和统计方法,走向最新的机器学习乃至深度学习算法,这一个个结构的神秘面纱正在逐步被我们揭开。
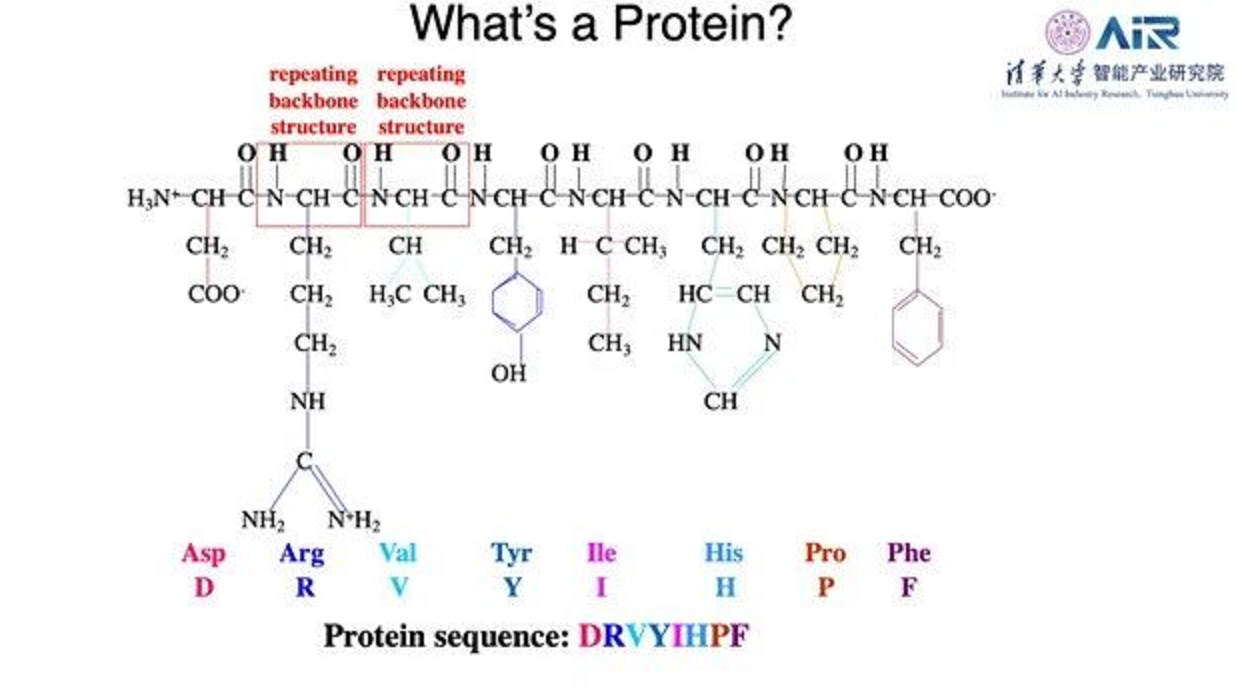
蛋白质是由一个个氨基酸按某种特定的顺序组成的。蛋白质序列经过折叠在空间中堆叠成三维几何体。各种氨基酸共有的结构是主链(Backbone),它们的侧链(SideChain)具有不同的结构。在蛋白质中一个氨基酸也被称为残基(Residue)。不同氨基酸的侧链往往具有不同的物理化学特性,例如亲水性、疏水性、大小不同,这些特性会影响氨基酸之间的组合关系,进而影响蛋白质结构。
预测蛋白质的结构有助于我们在原子层面了解蛋白质的功能。几十年来,人们一直在尝试着解开蛋白质结构的谜团,相应的算法也在不断更新。主要可以分为基于模板的和不基于模板的两类。但是挑战依然存在,因为一个蛋白质有可能会有上千个氨基酸,每个氨基酸有几十个原子。即使只考虑骨架,蛋白质结构也会有很大的自由度。机器学习算法的挑战在于预测新的折叠结构,也就是在训练数据里根本不存在的结构。深度学习需要大量训练数据,但是我们并没有那么多的实验技术解出来的蛋白结构。比如膜蛋白,对人体非常重要,但是相关实验结构数据却非常少。到2015年,不基于模板的预测成功率还是非常低。

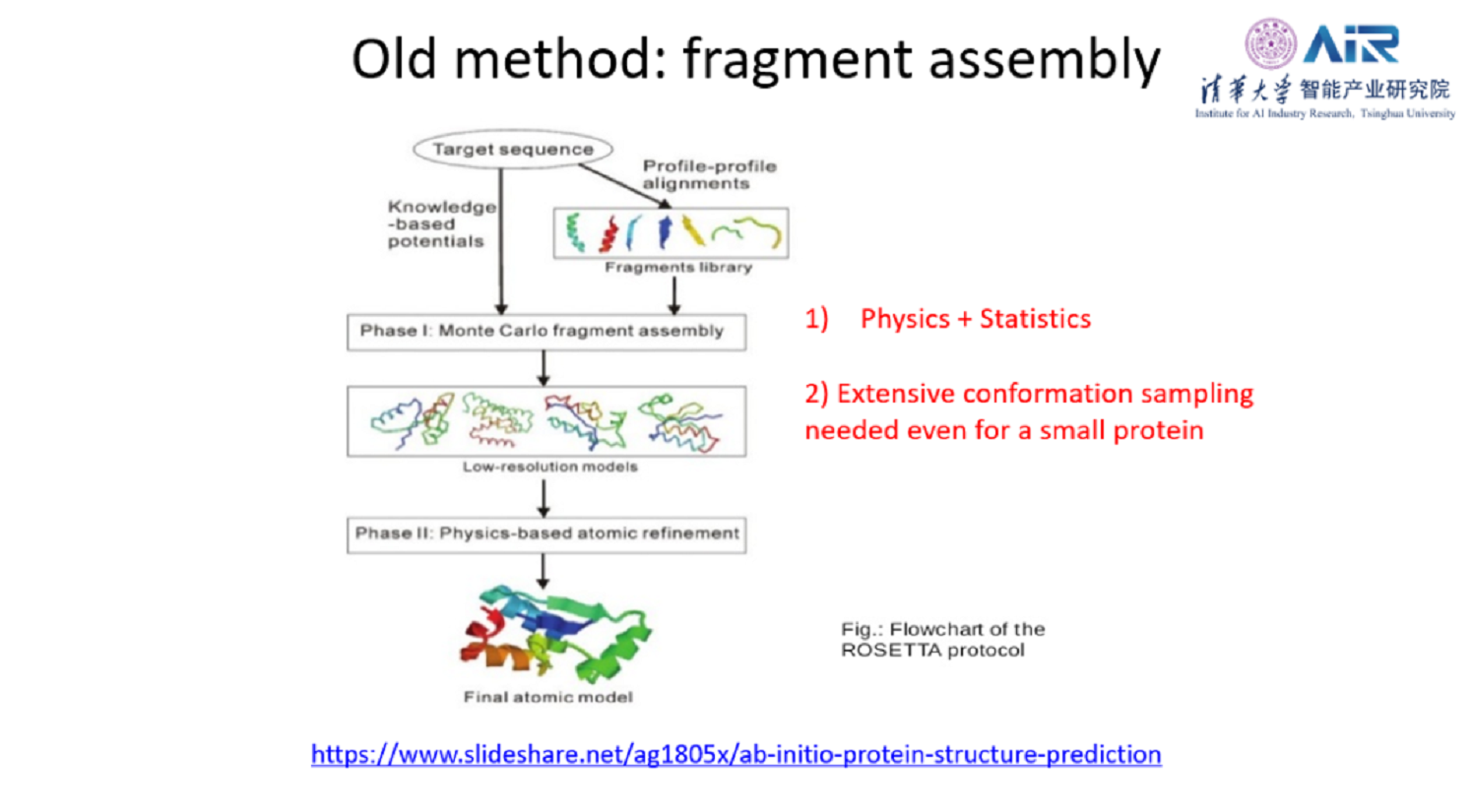
以前的方法从物理+统计的角度来解决这个问题。很流行的一种方法叫做片段拼接(fragment assembly),对一个蛋白序列,找不到同源结构的时候,可以把它分割成很多小的序列片段。每个小片段可以到结构数据库里去找相似的结构片段,然后通过采样把这些结构片段拼接起来形成一个完整的结构,最后计算能量函数去评估它的好坏。但是这种方法的问题在于大规模采样需要非常多的计算资源,一个很小的蛋白都要采样10万次,并且成功率很低。
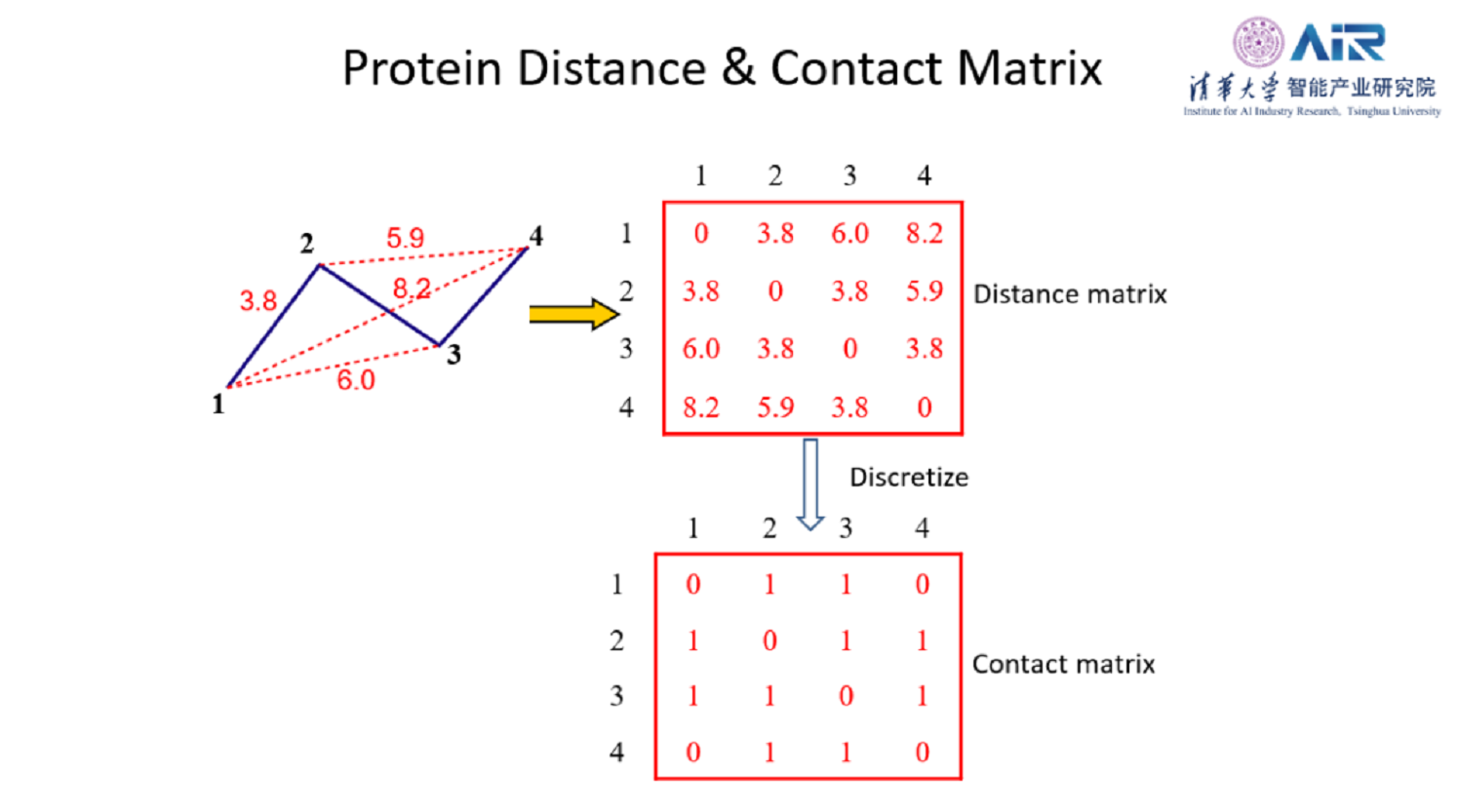
我们最近一些工作则提出不通过采样,而是基于预测氨基酸之间的关系去预测结构的想法。这种方法之前在九十年代就被提出,但是困于没有准确预测氨基酸之间关系的模型,一直没有广泛运用,直到2010年以来,才又受到关注。这种思想的背后是先用距离矩阵(distance matrix)和接触矩阵(contact matrix)去表示氨基酸之间的关系,之后的工作就是去准确地预测出接触矩阵。第一种成功的方法是基于一种共进化的概念。也就是当两个氨基酸离得近时,他们可能会共同突变和进化。
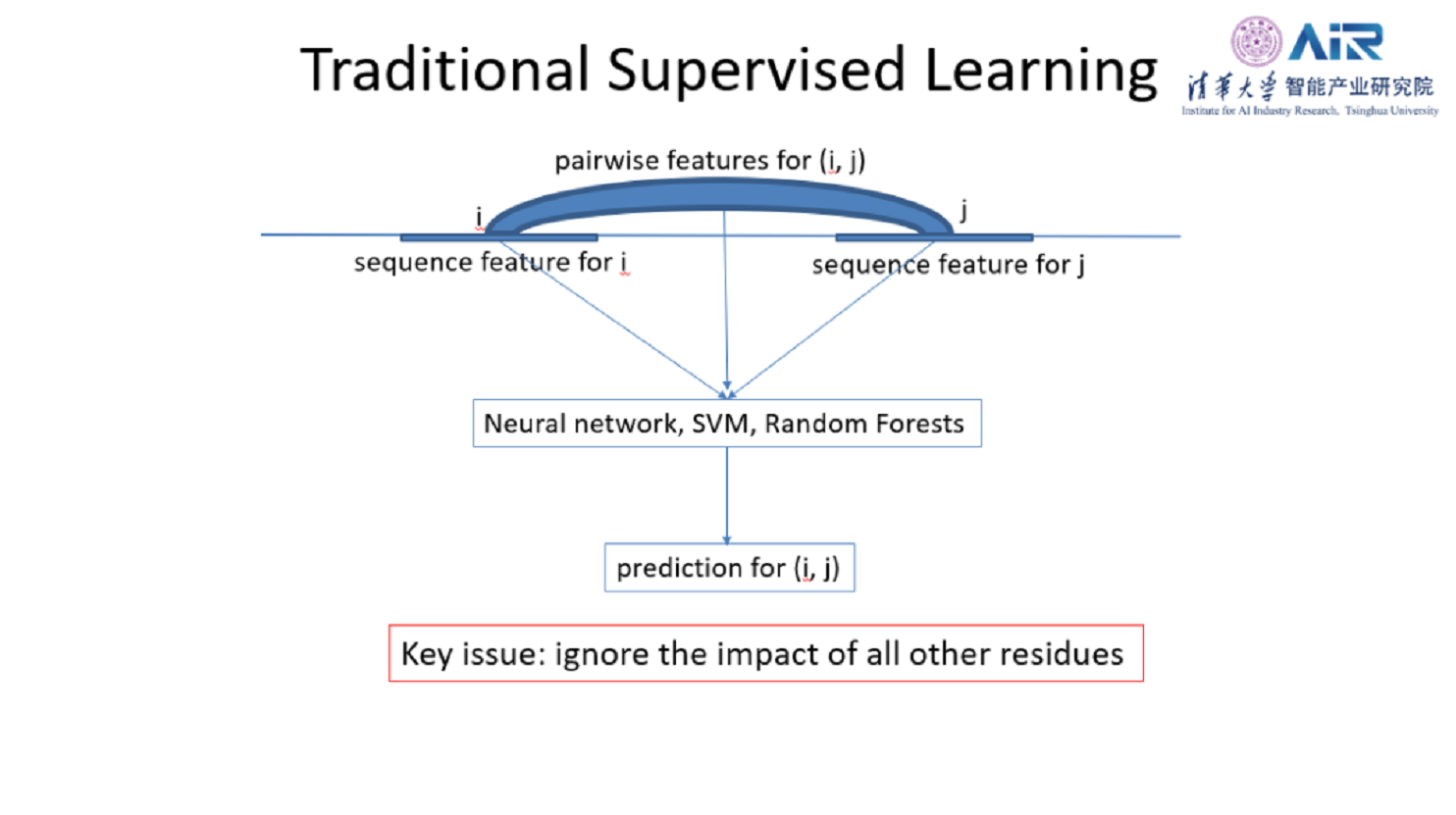
早期人们通过全局性的统计方法来预测共进化关系,但是这种方法局限性在于需要大量不冗余的同源蛋白序列。另一种方法是利用监督式学习,假设有一个蛋白质序列,要预测第i和第j个氨基酸有没有关系,可以把这两点的特征提取出来,用神经网络,SVM或者Random Forest去预测。这些方法在测试数据上一直不够精准。在2012年也有别的组尝试把机器学习换成深度学习,但是简单的替换对预测性能还是没有提升。
许教授接着提到自己在2016开始的工作,他们把接触矩阵看成是一个图片,每一个原子对都是看做一个像素(pixel),这样的话就可以同时预测所有pixel的label。这种方法借鉴了计算机视觉里语义分割的思路,并且使用了深度残差网络(ResNet)进行建模。
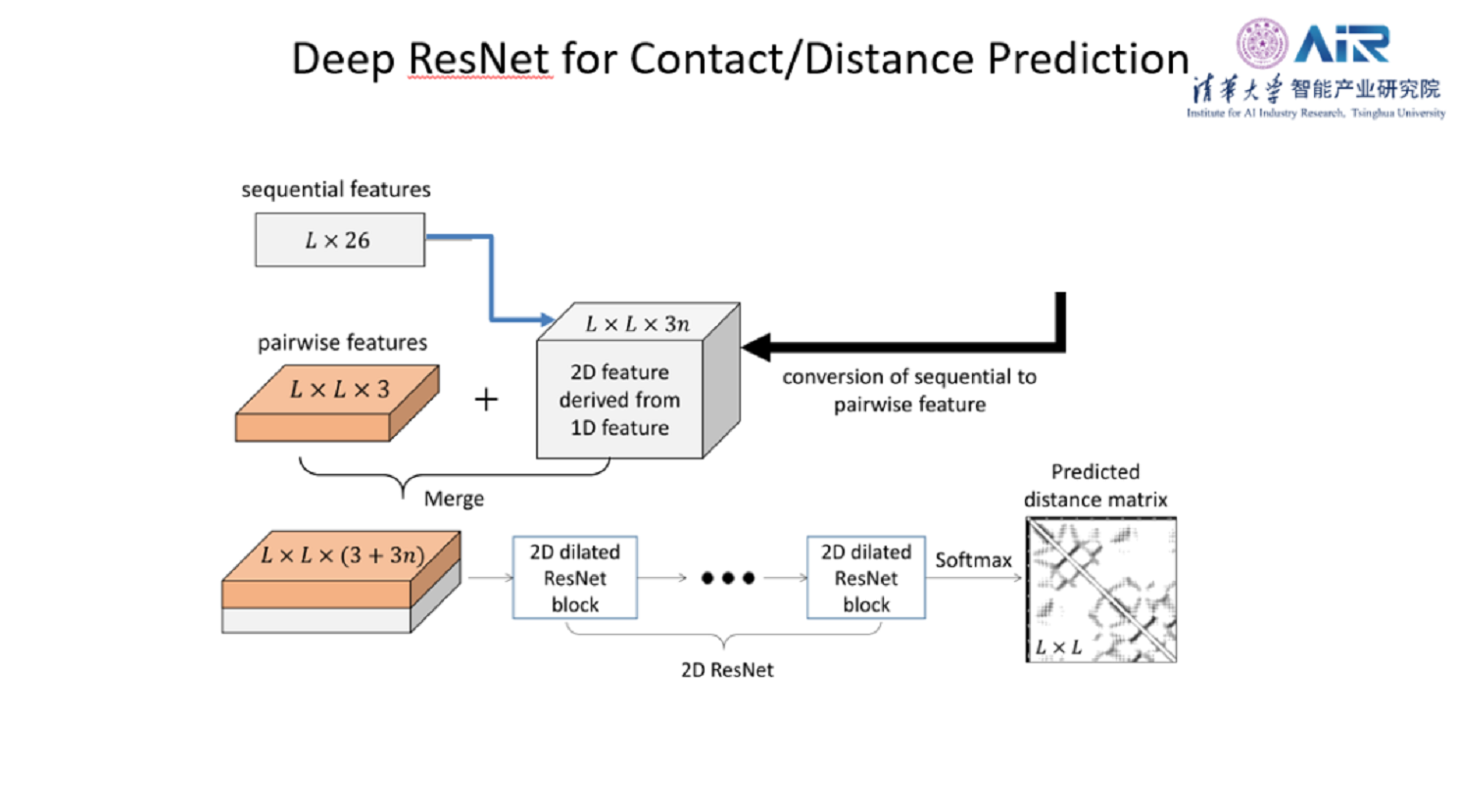
首先通过序列比对,找到同源序列,然后计算氨基酸的保守性,氨基酸之间的互惠信息(mutual information)和共进化信息。将这些一维和二维信息合并输入到残差网络中,就可以进行接触矩阵的预测。在这个架构图中二维信息要比一维信息重要的多,所以用的二维残差模块更多。在CASP12的比赛中,许教授团队用未完成的代码也取得了第一名的成绩。
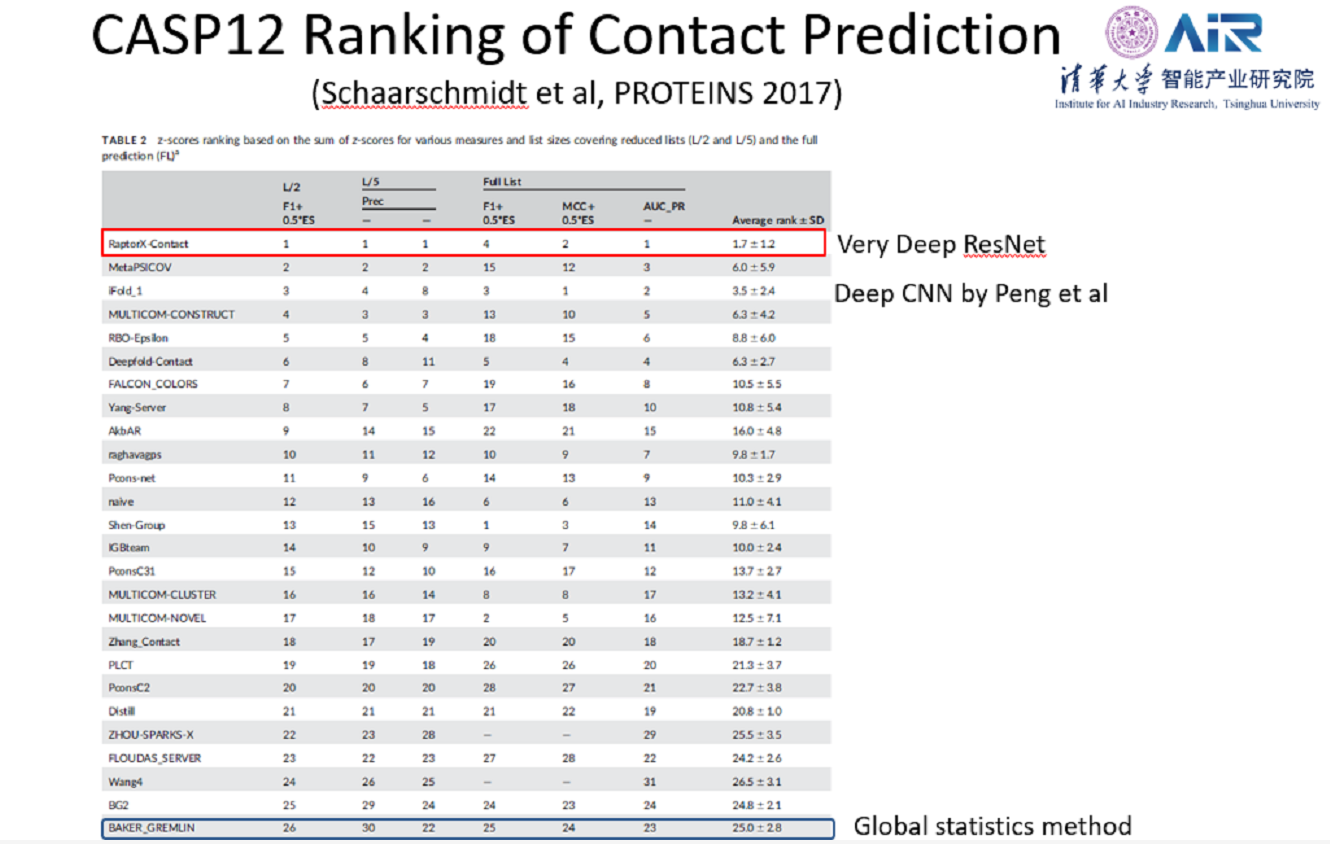
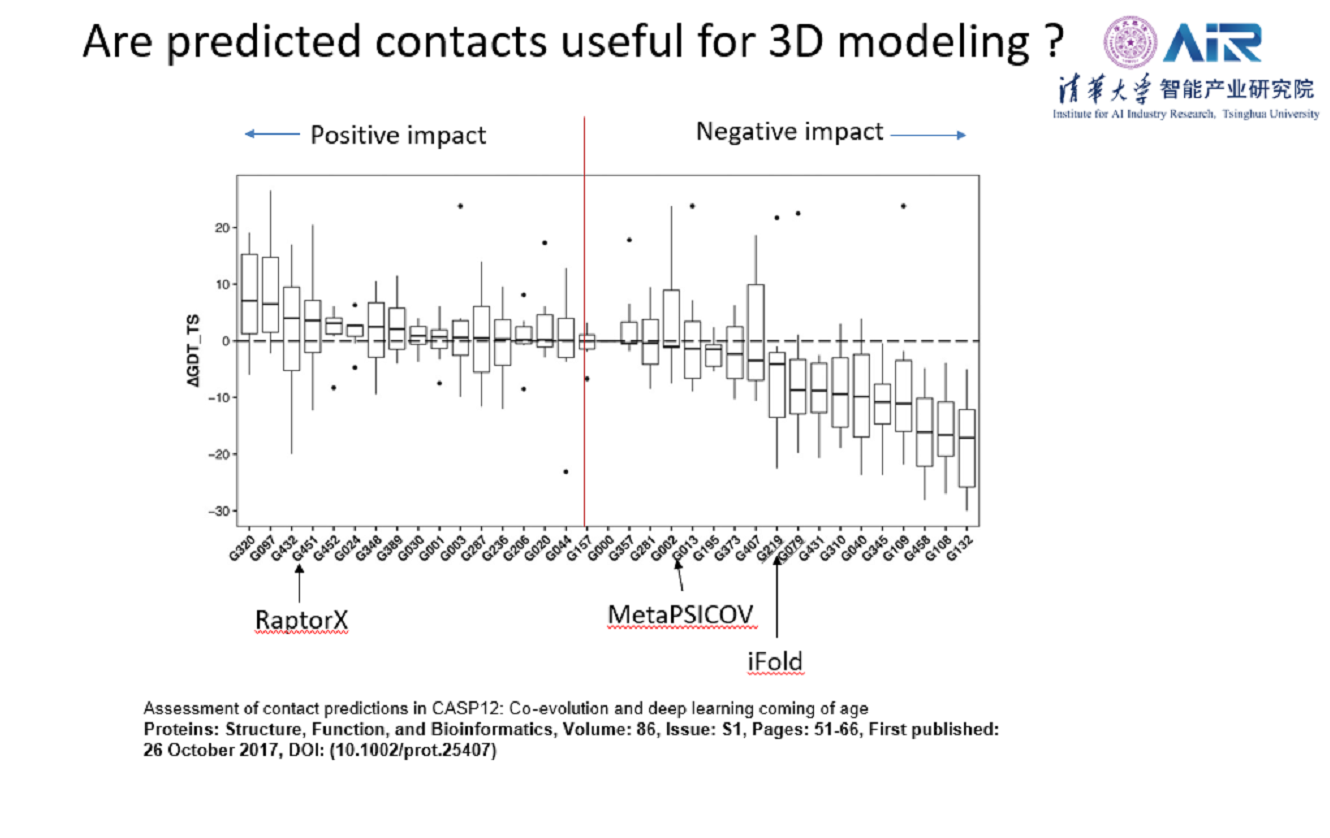
排在后面的如iFold也有用深度学习的,但是相对较浅,也没有残差模块,而许教授的残差模块堆叠到了60层。仅仅用这个接触矩阵的排名还不能说明预测出来的接触矩阵对3D建模有用,通过CASP评委公布的信息,发现许教授组的算法RaptorX对3D建模起到了非常正向的作用,而第二三名的算法却起到了负作用。
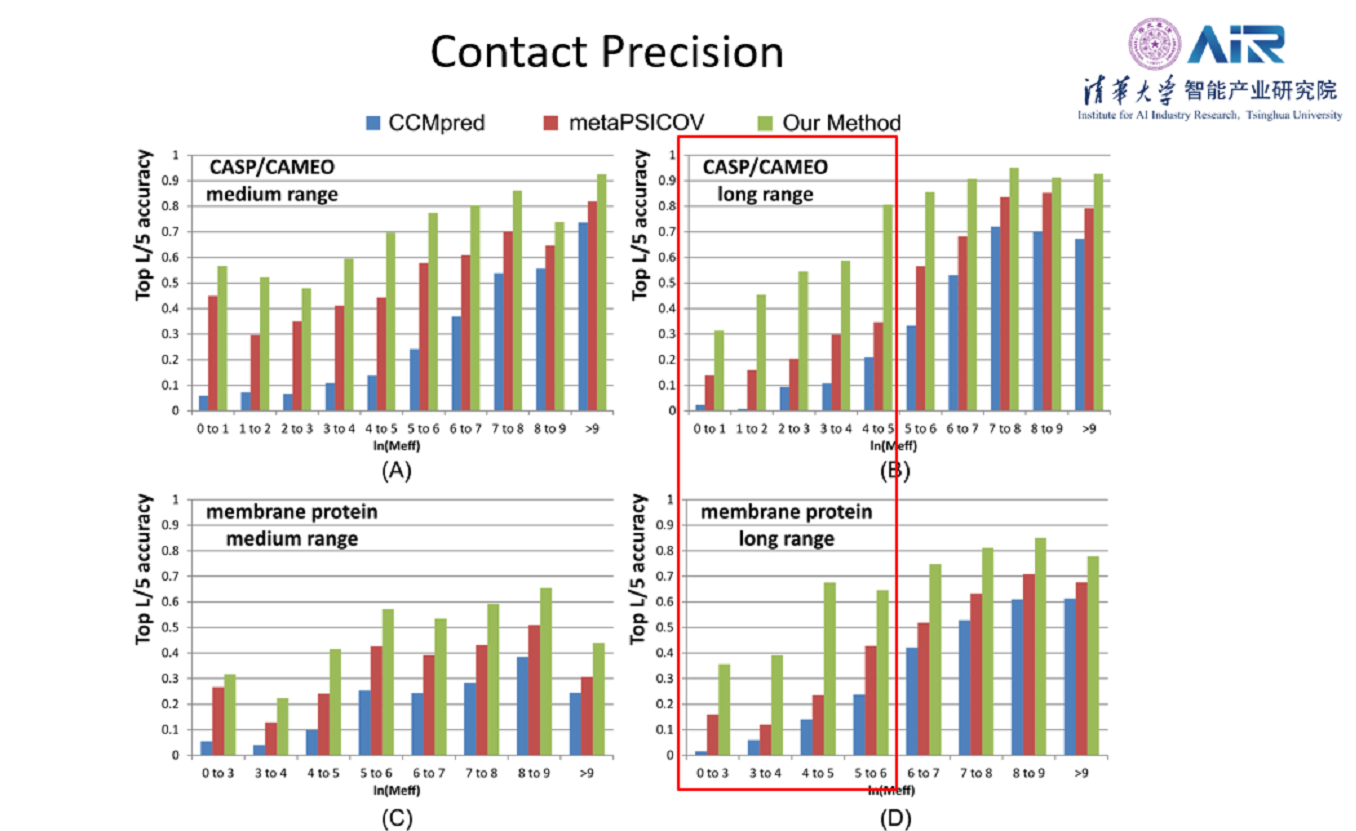
在CASP比赛之后,团队完成了完整版代码库,并且将文章发表在了PLOS Computational Biology上。后续基于全卷积的残差神经网络的工作也陆续发表在2017年的CellSystems,(膜蛋白预测相关工作)和2018年的NAR(两个蛋白之间的接触矩阵的预测相关工作)上。在CASP和CAMEO比赛数据集上,都取得了不错的效果,特别是在长程(longrange)接触的预测精度上更是远远好于CCMpred和metaPSICOV。
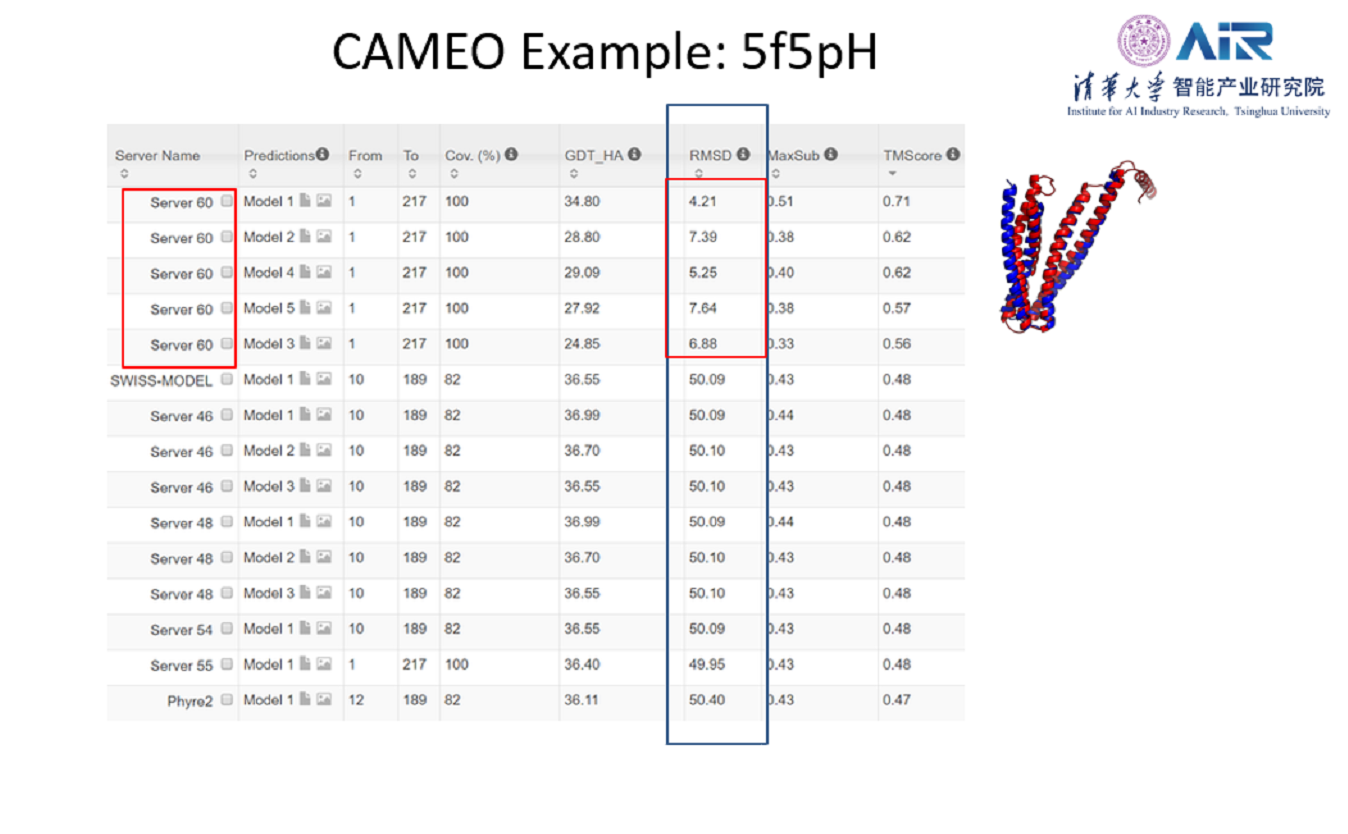
而且对后续3D建模的准确率提升也非常大。一个有趣的例子是在5f5pH测试蛋白上,许教授团队的算法在RMSD指标上远好于基于同源数据库预测的算法,原因在于其他服务器是通过搜索同源蛋白做的预测,然而搜索到的蛋白3thfA只有一部分相似,这就误导了其他服务器。

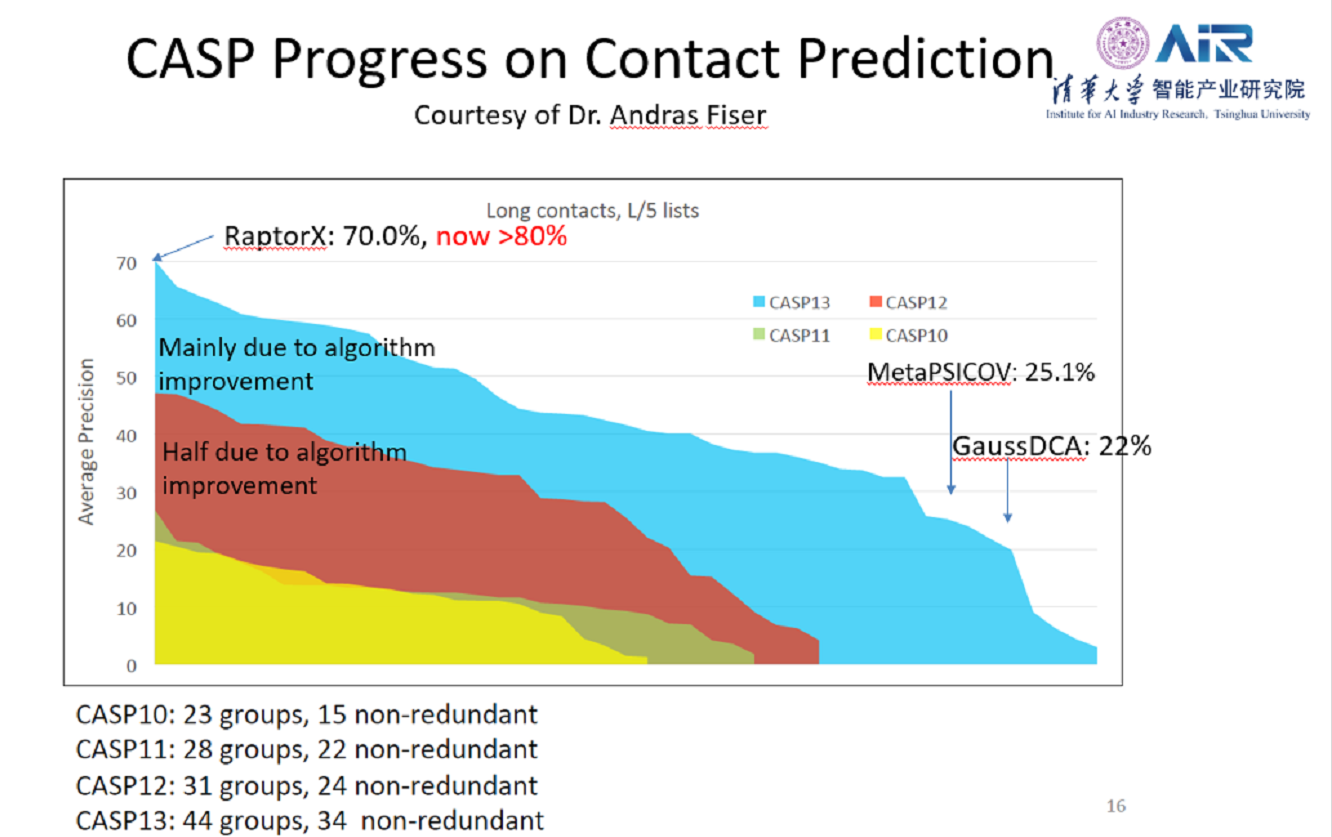
接着,许教授提到最近十年中CASP竞赛的一些变化,从CAPSP10到CASP13,最好的算法产生的结果在逐步提升,这主要是由于研究人员们提出了更加可靠的算法。
在3D建模方面,CASP13中Deepmind基于许教授的思想开发出了AlphaFold,效果好于其他的软件,但是在比赛之后,改进版RaptorX却能够达到超越AlphaFold的性能。
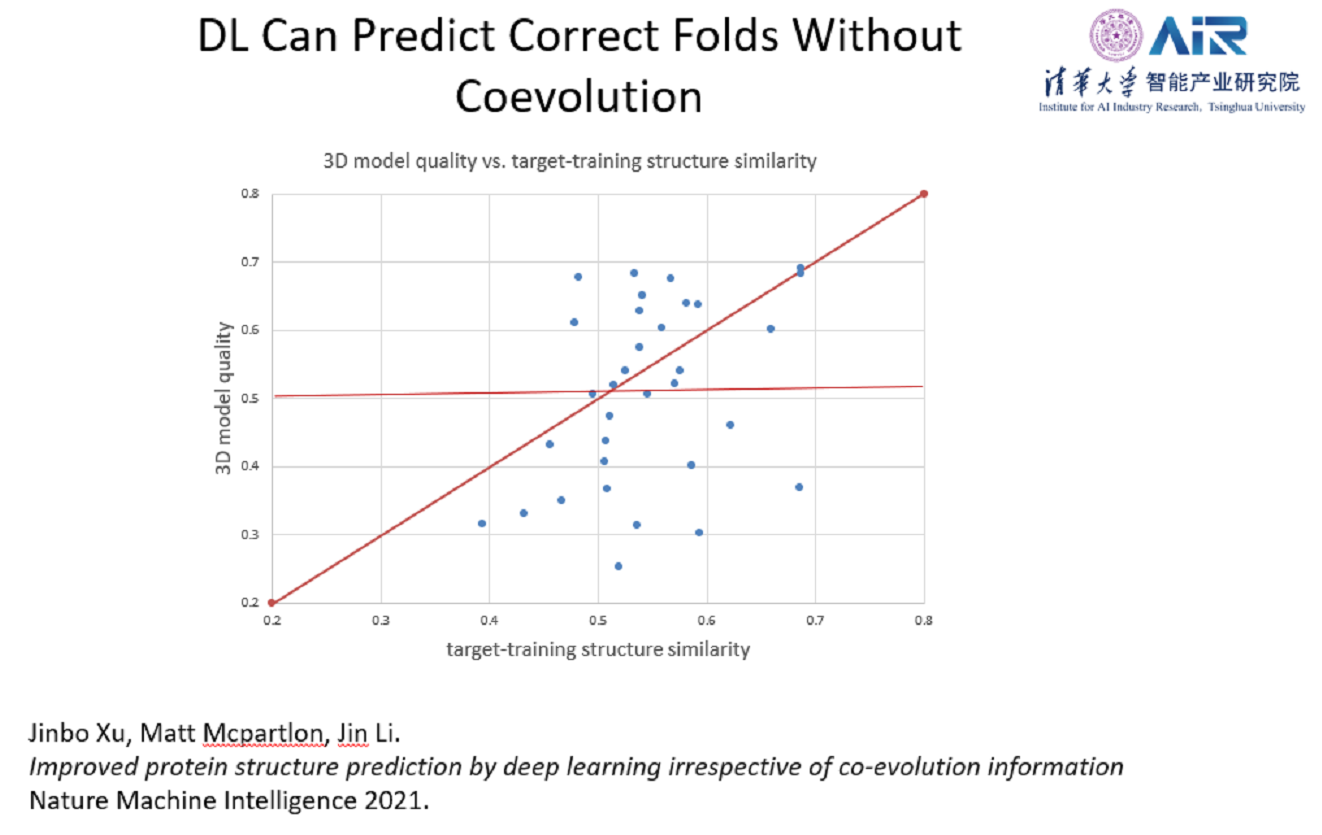
最近,许教授团队研究了在没有共进化信息的时候只用深度学习预测蛋白质结构能够达到的极限,发现了深度学习也比一般算法要好很多。并且,在人工设计的新蛋白方面,深度学习也可以把形状预测对,尽管精度不是很高,这也说明共进化信息不是深度学习中必须要用的特征。
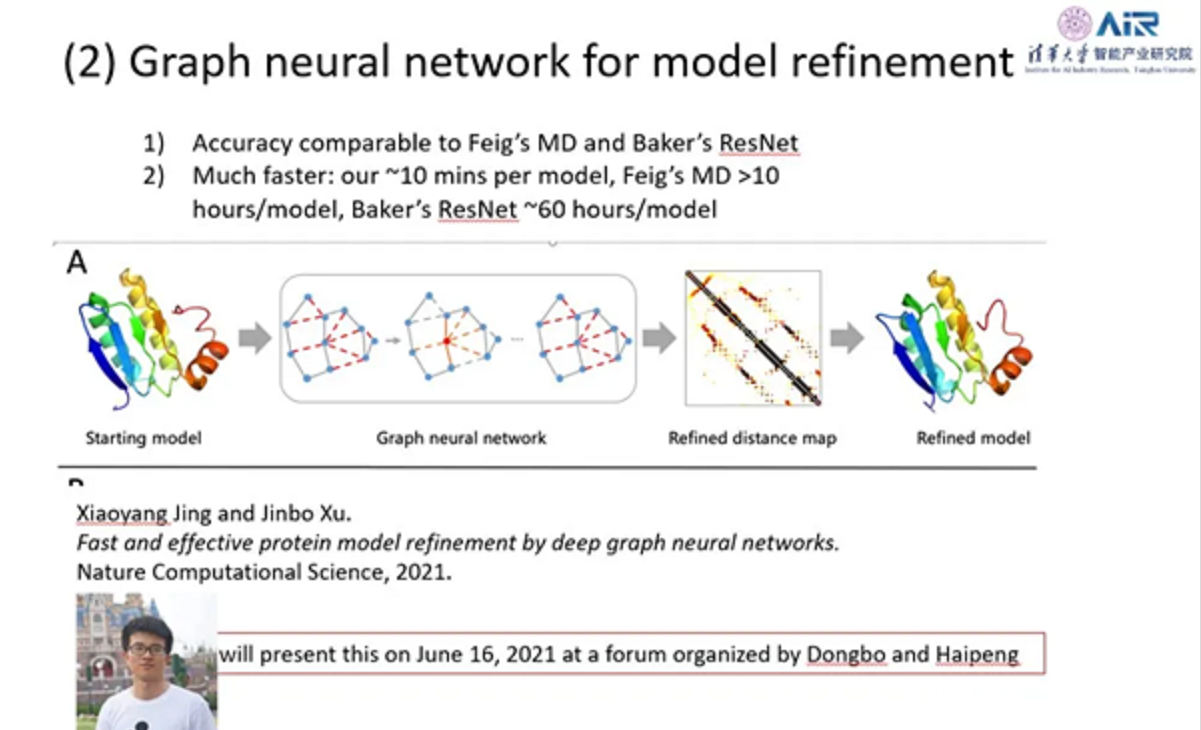
最后,许教授展望未来蛋白质结构预测方面的进展,提到很多组都有很多新的进展。比如Facebook最早提出可以更好地利用序列和结构信息,用Transformer进行无监督学习,而Deepmind更是做到了端到端的优化,成功通过监督学习达到了空前的效果。包括许教授团队,Deepmind,Baker在内的团队也同时在考虑引入模板信息到深度学习中去,这对某些特别大的蛋白结构预测非常重要。Kentaro Tomii, DongboBu等学者发现从MSA中学习序列权重对于最后的结构预测有正向作用。此外,基于最近图神经网络(GNN)的发展,用GNN进行蛋白结构模型进行优化也是许教授团队最近研究的课题。GNN跟基于分子动力学的方法的效果接近,但是比基于分子动力学的方法快很多倍。
关于模板的引入,可以发现在只用深度学习没有模板的情况下,单个domain的结构都可以预测准确,但是完整的结构却并没有做对。在只有模板没有深度学习的情况下,可以发现整个结构框架基本可以做对,但是单个domain的结构却不是很准确。而在同时结合了深度学习和模板信息后,既可以准确预测单个domain的结构,又可以成功地预测完整的三维结构。
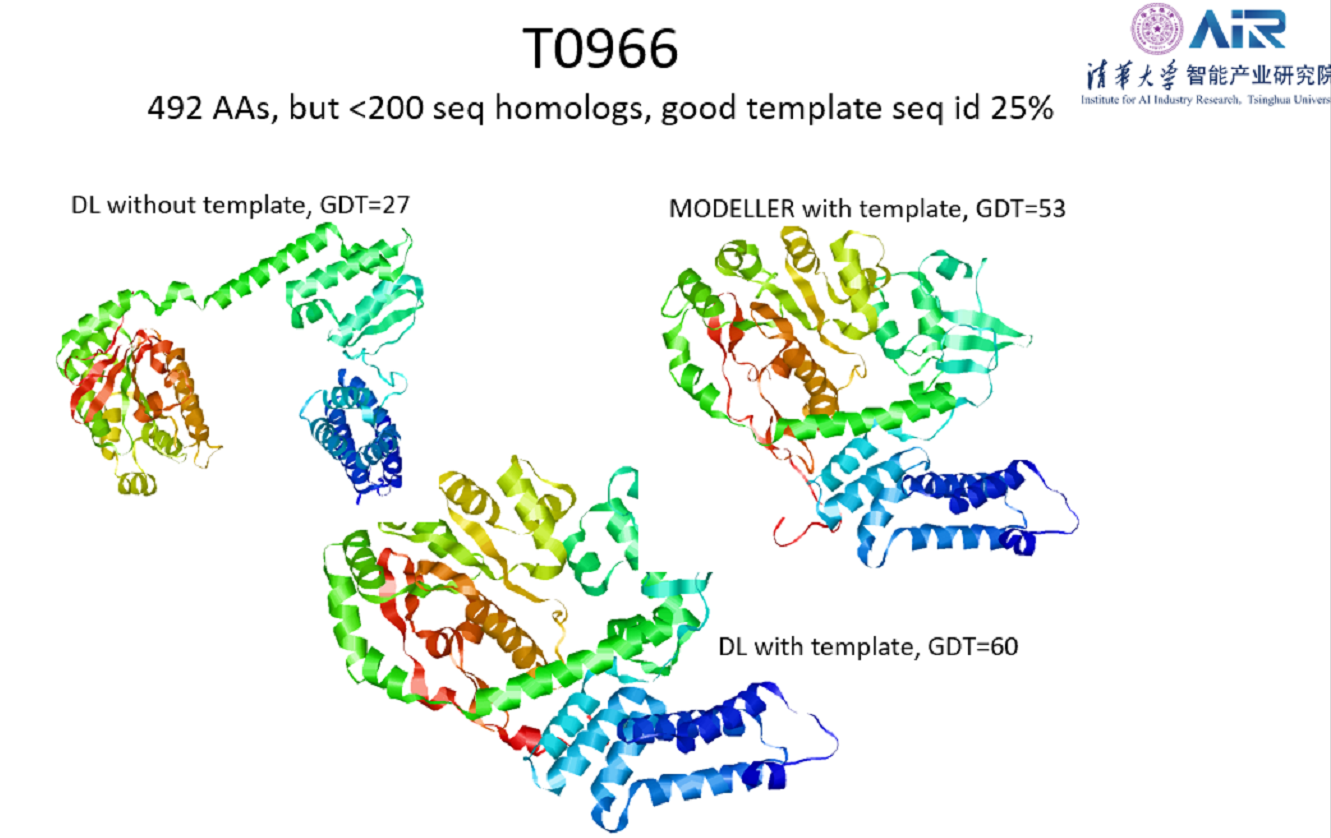
为了更好地方便热衷于蛋白质结构预测的同学们入行,许教授最后简单介绍了CASP比赛的一些信息和最新的AlphaFold2模型自己的理解。通过CASP比赛的对比图,可以发现对于最难的蛋白,在CASP13之前基本没有提升,因为没有那么多序列让模型进行共进化分析,而真正有效的提升还是基于深度学习的方法,尤其是许教授提出的残差网络和AlphaFold2的Transformer。
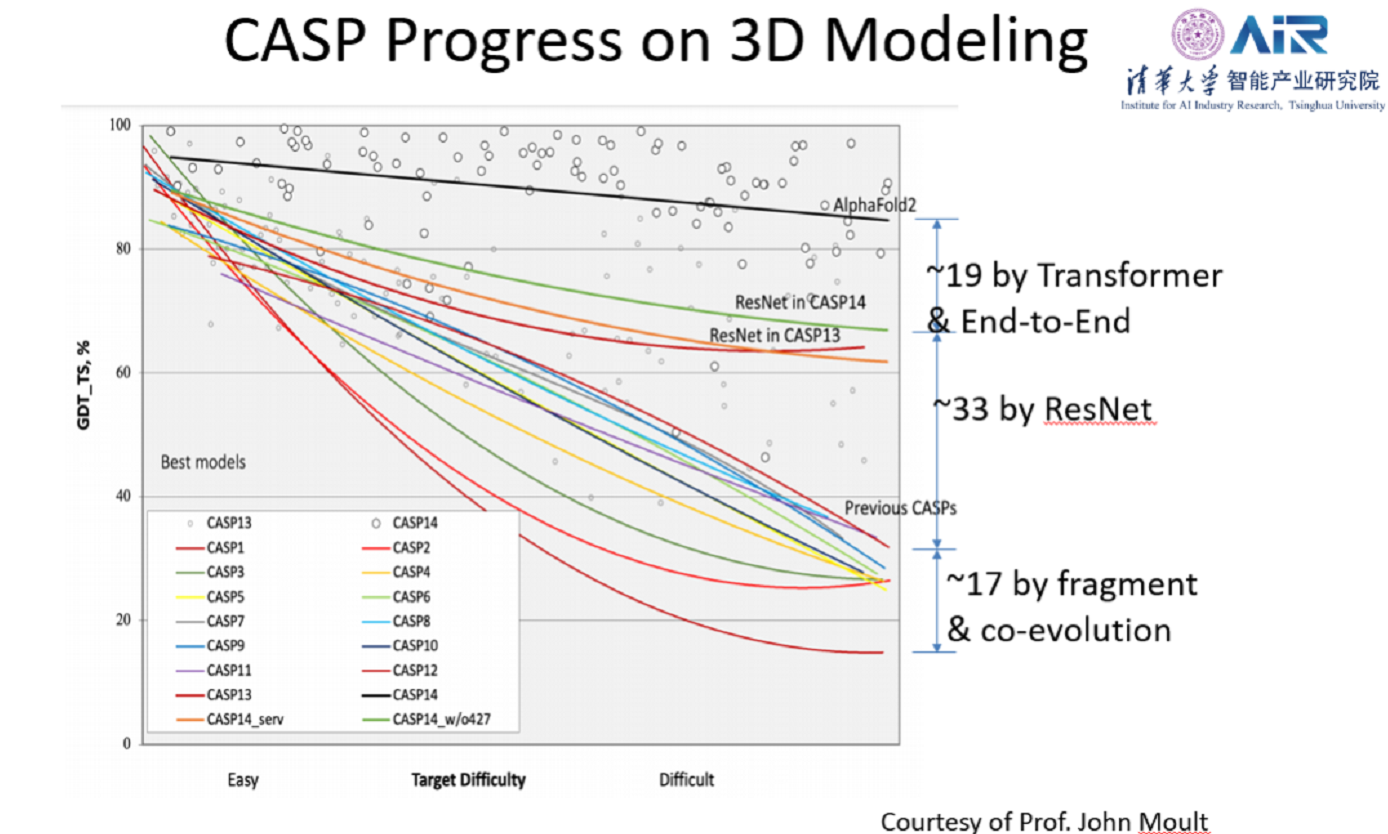
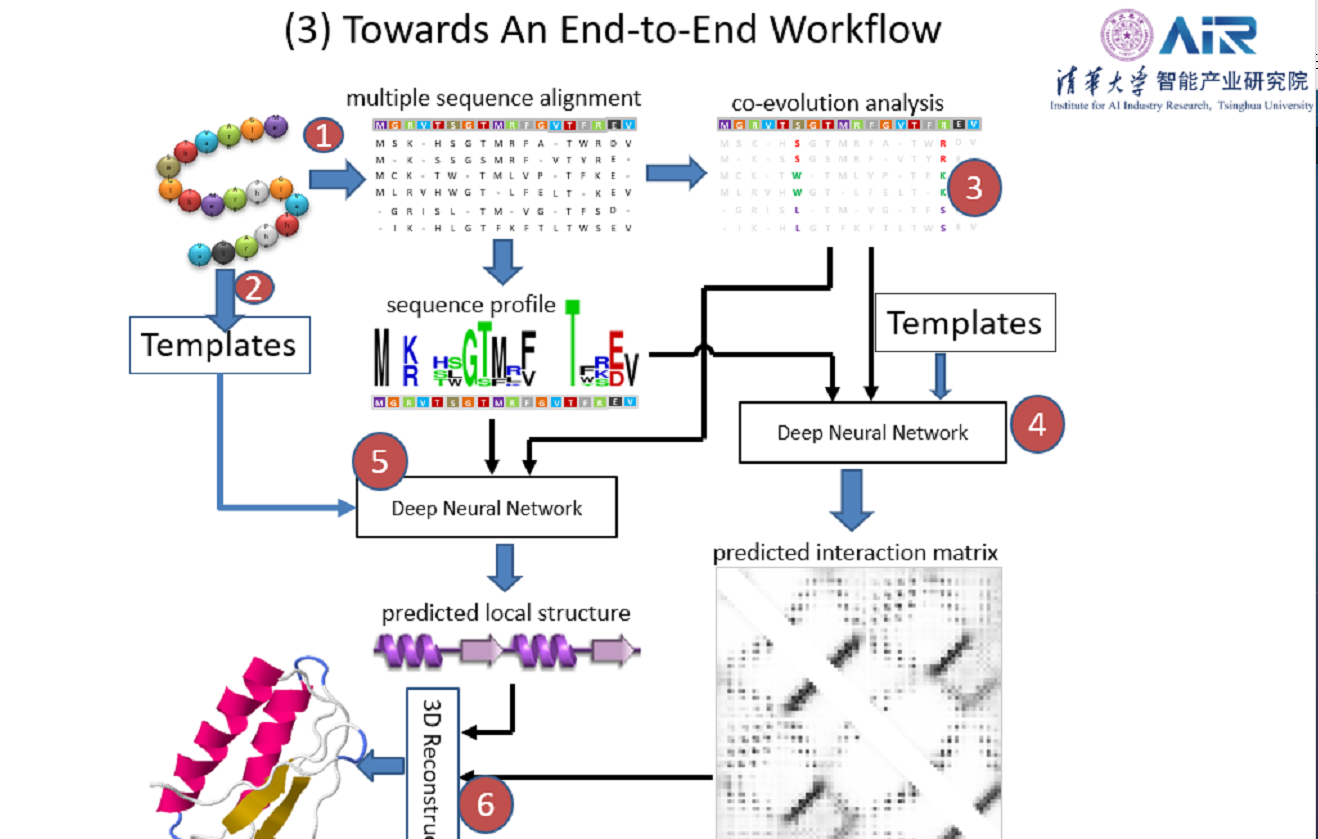
可见深度学习对于结构预测起到了很大的推动作用。从图中还可以看见AlphaFold2基本在CASP14中一枝独秀。鉴于还没有公布论文和代码,许教授对于这个模型给出了自己的见解。在许教授的方法中,要去先搜集同源序列,堆在一起得到MSA,同时也找一些模版,通过MSA可以输入神经网络预测氨基酸相互作用进行共进化分析。同时,也用另一个神经网络进行预测局部结构和二级结构。最后在把这些预测的结构信息输入一个优化模块去预测三级结构。可以看见这个模型有六个模块,每一步都是独立的,都是分开来做的,后面步骤的误差没法反馈到前面的模块。
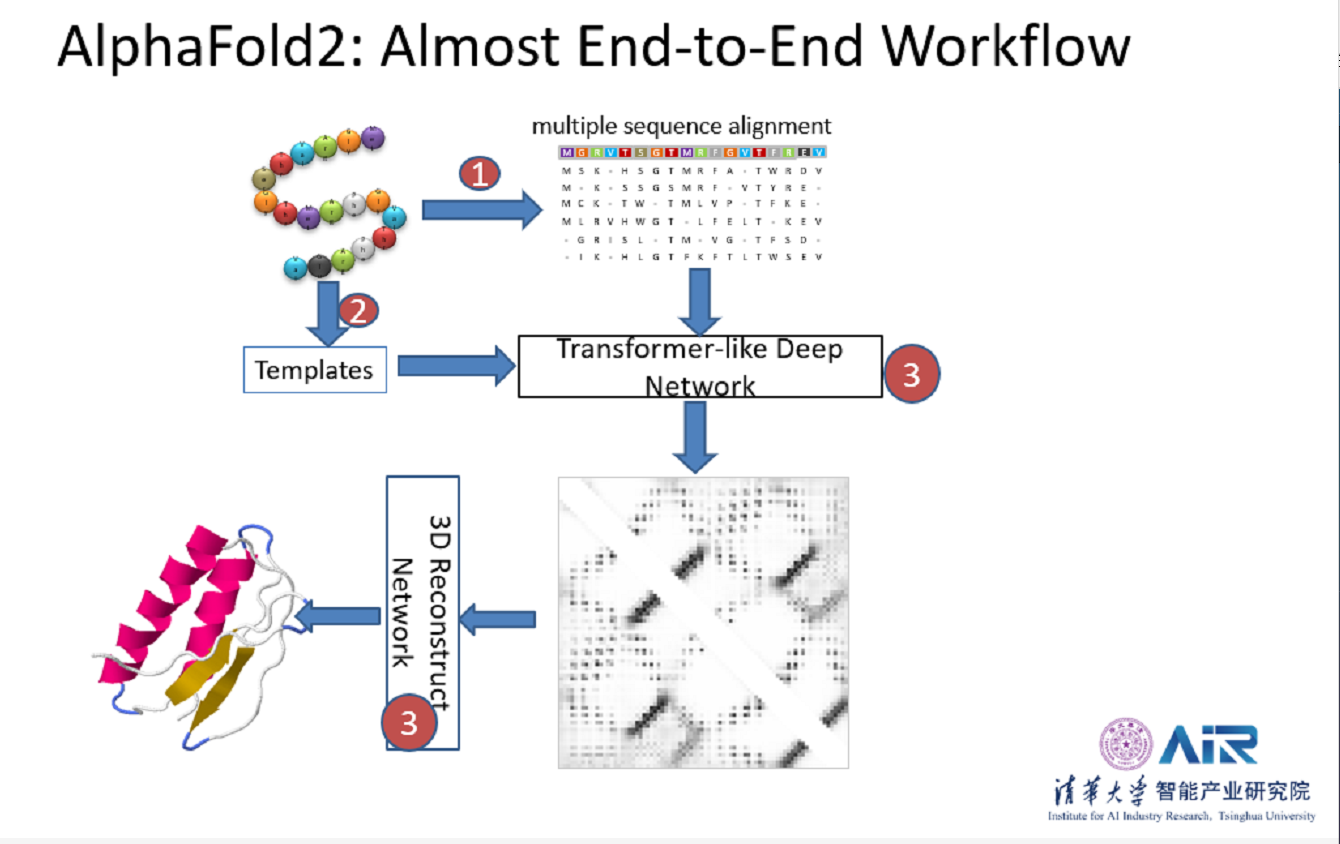
而AlphaFold2基本是一个端到端的方法,找到同源序列后输入到一个神经网络,预测两个氨基酸之间的相互关系,然后去建立一个三维模型来预测三级结构。这个模型只有三步,第一步是找同源序列,第二步是找模版,第三步是靠氨基酸之间的关系把原子坐标预测出来。这样目标就简单了,基本是端到端的预测流程,误差可以反馈到前面Transformer网络里面去,提高预测精度。当然他们的模型效果好的原因不仅仅是用了Transformer,还用了端到端的实现,还雇佣了专门的专家去搜索同源序列。因为AlphaFold2比以前的方法有非常多的改进,并且并未公开论文和代码,所以具体哪一个模块使得AlphaFold2的效果如此之好还是值得探索。
那么CASP是怎么组织的呢?参赛者分为服务器组和人工组两种,服务器组预测一个蛋白质结构只有三天时间,并且看不到其他服务器的结果。人工组则有三周时间,并且可以看到和使用所有服务器提交的结果。所以这样的话人工组可以通过简单地集成服务器组的结果来进行预测,这样的人工组的结果会比大部分服务器组好,但是没有太多的学术价值。服务器组也可以用类似的方法,也就是通过下载和集成各种各样的预测软件来提高性能。另外使用越来越大的蛋白质序列数据库也是很重要的。
在报告最后,许教授对蛋白质结构预测这个任务的发展进行了总结。经过这几年的研究者们的探索,发现深度学习对蛋白质结构预测的提升效果是有显著效果的,可以把大部分蛋白质都三维空间大致形状预测准确。而最近的Transformer模型和端到端训练的引入使得这项任务的准确性又上了一个高度。许教授还强调,在这项任务中,非常重要的思想是要对整个蛋白建模,而不是取出两三个氨基酸单独建模。通过深度学习,现在人们已经可以预测各种规模的蛋白质结构,而且模型运行速度也很快,蛋白质结构预测这个任务成为了广大研究者都可以探索的问题,很多组都可以在本地跑算法。一旦模型训练好之后,我们甚至可以在自己的笔记本上测试运行。对于想要快速入门的同学,许教授也提到在这个领域最近发表的两篇综述论文,方便同学们了解这个领域最新的进展。

最后,许教授对所有参与工作的学生和同事表示了感谢,在掌声中结束了演讲。
撰文 / 丁凡 刘聆羽
编辑排版 / 刘聆羽
校对责编 / 黄妍
精彩视频回顾及完整版PPT下载,请点击:
AIR学术沙龙第5期|许锦波:基于深度学习的蛋白质结构预测


