清华大学智能产业研究院(AIR)马维英教授、聂再清教授、周浩副教授课题组与北京大学、南京大学以及水木分子的研究团队提出了 ESM-AA 模型。该模型在蛋白质语言建模领域取得了重要进展,提供了一套整合多尺度信息的统一建模方案。它是首个能同时处理氨基酸信息和原子信息的蛋白质预训练语言模型。模型的出色性能展示了多尺度统一建模在克服现有局限和解锁新能力方面的巨大潜力。作为基座模型,ESM-AA 获得了多位学者的关注与广泛讨论,被认为有潜力基于 ESM-AA 开发出可与 AlphaFold3、RoseTTAFold All-Atom 相竞争的模型,为研究不同生物结构间的相互作用开辟了新的道路。当前论文已被 ICML 2024 录⽤。

· 论文标题:ESM All-Atom: Multi-Scale Protein Language Model for Unified Molecular Modeling
· Github开源地址:
https://github.com/zhengkangjie/ESM-AA
· 论文链接:https://arxiv.org/abs/2403.12995
蛋白质是各种生命活动的关键执行者。深入理解蛋白质及其与其他生物结构的相互作用是生物科学中的核心议题,这对靶向药物筛选、酶工程等领域具有显著的实际意义。
因此,如何更好地理解与建模蛋白质也成为了目前 AI4Science 领域的一个研究热点。
近日来,包括 Deepmind、华盛顿大学 Baker 组在内的各大前沿研究机构也针对蛋白质全原子建模问题展开了深入研究,提出了包括 AlphaFold 3、RoseTTAFold All-Atom 等针对蛋白质以及其他生命活动相关分子的全原子尺度建模模型,可以在很高的精度下实现对蛋白质结构、分子结构以及受体-配体结构等全原子尺度的精确预测。
虽然这些模型对于全原子尺度的结构建模取得了重大进展,目前主流的蛋白语言模型仍然无法实现全原子尺度的蛋白质理解与表示学习。
多尺度,下一代蛋白质模型的「必经之路」
以 ESM-2为代表的蛋白质表示学习模型,它们以氨基酸作为构建模型的唯一尺度,这对于专注于处理蛋白质的情境而言是一种合理的方法。
然而,要全面理解蛋白质的本质,关键在于阐述它们与其他生物结构(如小分子、DNA、RNA 等)之间的相互作用。
面对这种需求,需要描述不同结构间复杂的相互作用,单一尺度的建模策略难以提供有效的全面覆盖。
为了克服这一缺陷,蛋白质模型正在经历一场向多尺度模型转变的深刻革新。例如,5月初发表在《Science》杂志上的 RoseTTAFold All-Atom 模型,作为 RoseTTAFold 的后续产品,引入了多尺度概念。
这一模型不仅仅局限于蛋白质结构预测,还拓展到了蛋白质与分子/核酸的对接、蛋白翻译后修饰等更广泛的研究领域。
同时,DeepMind 最新发布的 AlphaFold3 也采用了多尺度建模策略,支持预测多种蛋白复合物的结构,其表现令人瞩目,无疑将对人工智能和生物学领域产生重大影响。
ESM All-Atom
多尺度的蛋白质语言模型基座
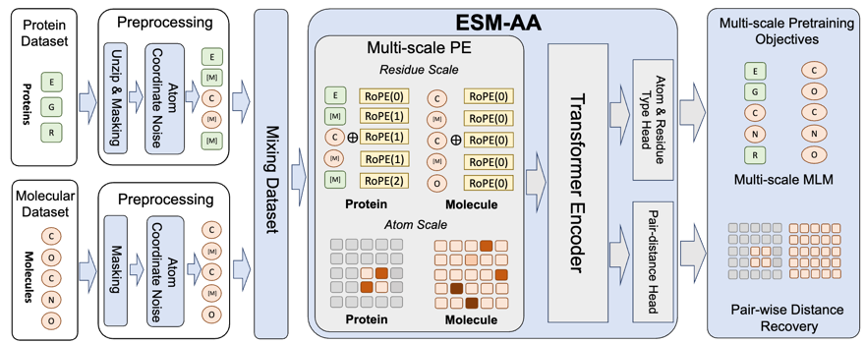
RoseTTAFold All-Atom 和 AlphaFold3 对多尺度概念的成功应用启发了一个重要思考,即:作为蛋白质基座模型的蛋白语言模型应如何采纳多尺度技术。基于此,该团队提出了多尺度蛋白质语言模型 ESM All-Atom(ESM-AA)。
简要来说,ESM-AA 通过将部分氨基酸「展开」(Unzip)为对应的原子组成引入了多尺度概念。随后,通过混合蛋白数据与分子数据进行预训练,这使得模型具备了同时处理不同尺度生物结构的能力。
此外,为了帮助模型更好地学习优质的原子尺度信息,ESM-AA 还会利用原子尺度的分子结构数据进行训练。而且通过引入图 2 所示的多尺度位置编码机制,ESM-AA 模型可以很好地对不同尺度的信息进行区分,确保模型能够精确理解残基层面与原子层面的位置与结构信息。
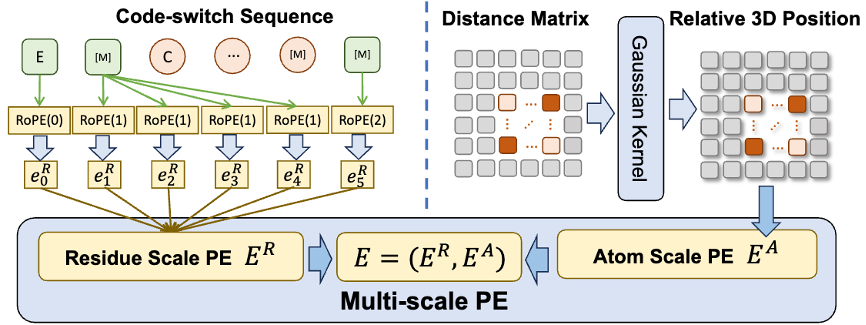
图 2:多尺度位置编码
多尺度预训练目标
为了帮助模型学习多尺度信息,该团队为 ESM-AA 模型设计了多种预训练目标。ESM-AA 的多尺度预训练目标包括掩码语言建模(MLM)和成对距离恢复(PDR)。如图 3(a) 所示,MLM 通过遮盖氨基酸和原子,要求模型根据周围的上下文进行预测,这一训练任务可以在氨基酸和原子两个尺度上进行。而 PDR 则要求模型准确预测不同原子之间的欧几里得距离,以训练模型理解原子级的结构信息(如图 3(b) 所示)。
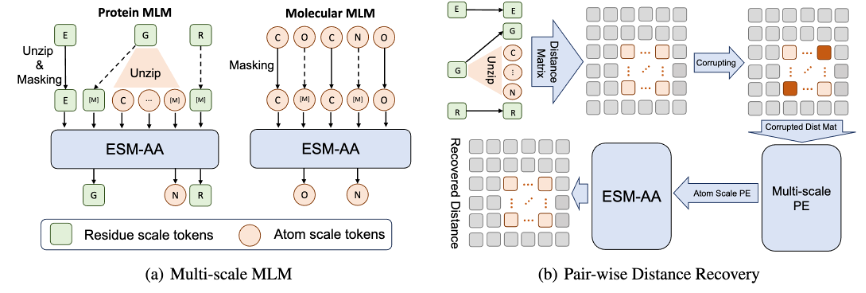
图 3:多尺度预训练任务
性能评估
ESM-AA 模型在多种蛋白-小分子基准任务上进行微调和评估,包括酶-底物亲和力回归任务(结果展示于图 4)、酶-底物对分类任务(结果展示于图 4)和药物-靶标亲和力回归任务(结果展示于图 5)。
结果显示,ESM-AA 在这些任务中优于之前的模型,表明其在氨基酸和原子尺度上充分发挥了蛋白质预训练语言模型的潜力。
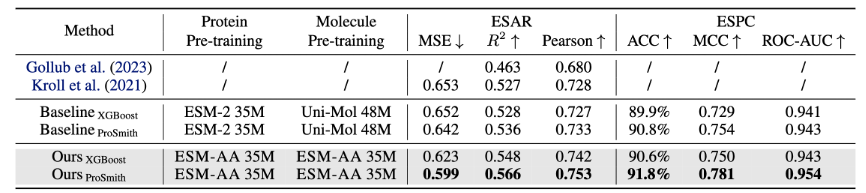
图 4:酶-底物亲和力回归任务以及酶-底物对分类任务性能比较
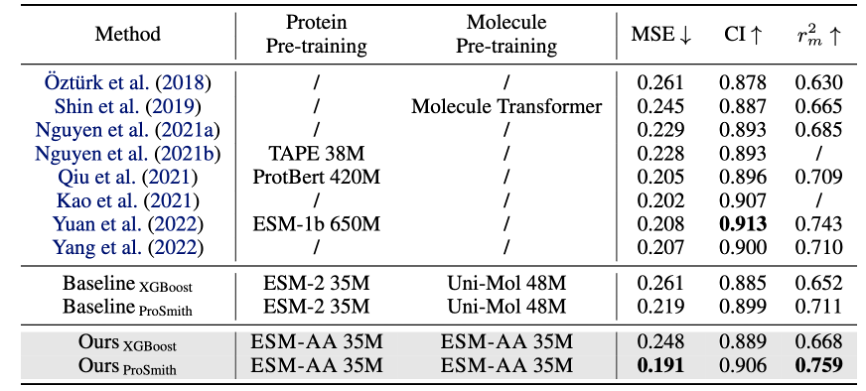
此外,ESM-AA 模型也在蛋白质接触预测、蛋白功能分类以及分子性质预测等任务上测试了性能。
结果显示,在处理仅涉及蛋白质的任务时,ESM-AA 的表现与 ESM-2 相当;在分子任务上,ESM-AA 模型的性能优于大多数基准模型,与 Uni-Mol 的表现相近。
这表明,ESM-AA 在获取强大分子知识的过程中并未牺牲对蛋白质的理解能力,也进一步说明了 ESM-AA 模型成功复用了 ESM-2 模型的知识,而无需被从头开始重新开发,显著降低了模型训练成本。
可视化分析
为了进一步分析 ESM-AA 在蛋白-小分子基准任务上表现优异的原因,该论文展示了 ESM-AA 模型以及 ESM-2+Uni-Mol 模型组合在该任务中抽取的样本表征分布的可视化结果。
如图 6 所示,ESM-AA 模型所学习的蛋白和小分子表示之间更为紧凑,这预示着两者处于同一表示空间,这是 ESM-AA 模型优于 ESM-2+Uni-Mol 模型的原因,进一步说明了多尺度统一分子建模的优势。

清华 AIR 团队开发的 ESM-AA 是首个融合氨基酸与原子信息处理的蛋白质预训练语言模型。模型通过整合多尺度信息,展现出稳健且卓越的性能,为解决生物结构间相互作用难题提供了新途径。
ESM-AA不仅促进了对蛋白质更深层次的理解,还在多项生物分子任务中表现出色,证明了其在保持蛋白质理解能力的同时可以有效融合分子层面知识,降低了模型训练的成本,为 AI 辅助的生物科学研究开辟了新方向。


