在生命科学领域,生成式AI已经展现出巨大的潜力和价值。生成式AI已经被用于药物发现和生物医学研究,它可以通过模拟生物分子的结构,预测新的药物分子或者预测蛋白质的三维结构。
10月14日,第5期AIR学术工作坊下半场的嘉宾:
智源健康计算研究中心负责人叶启威,为我们做了题为《AI for LifeScience》的报告。
叶启威,智源研究院健康计算研究中心负责人,AI for Science青年学者,曾任微软亚洲研究院主管研究员,研究方向包括(深度)强化学习,决策树模型,生成模型及其应用。在2016研发了LightGBM,在精度和速度上都超过同时代其他框架,成为业界最受欢迎的决策树算法之一。在2018年研发了Suphx,目前最强的麻将AI模型,在『天凤』平台荣升十段,显著超越人类顶尖选手。2023年其开发的OpenComplex蛋白质复合结构预测模型荣获CAMEO年度冠军。
叶启威首先介绍了他们的团队智源健康研究中心。
该团队由一群年轻的AI背景同学和研究人员组成,他们认识到AI在这个领域中存在巨大的机遇和挑战。
为了处理各种与分子相关的数据,他们需要开发新型工具和研究方案。
团队的目标是研发一些AI工具,希望最终能够为这个社区带来革命性的改变。
接着,叶启威讲到了生成式AI,并表示生成式AI已经在各个领域产生了重大影响。例如,我们熟知的ChatGPT,现在已经可以轻松地用于写文章、作诗。在计算图形学领域,我们已经看到了一些有趣的应用,如Stable Diffusion和OpenAI的Dalle。而今年,我们也看到了在音乐、视频和3D头像等领域涌现出越来越多的生成式模型。那么我们能否利用这些模型在生物分子领域构建一些有价值的应用呢?
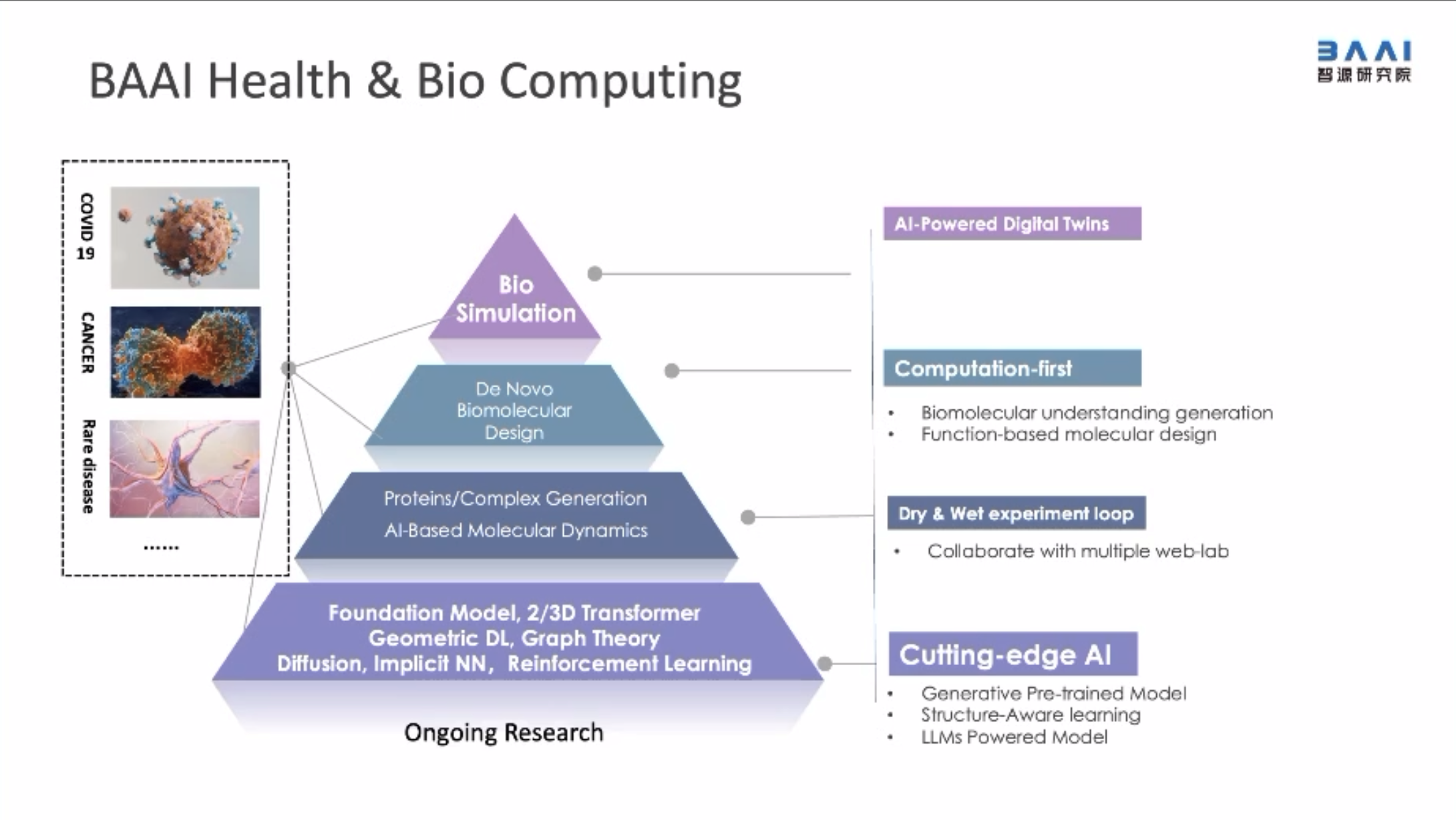
叶启威认为,为了实现在生物分子领域的ChatGPT时刻,我们需要构建一个适用的基础模型。这个基础模型需要完成三件事。首先,我们需要构建更好的生物分子表示方法。其次,我们需要构建结构理解模型,以揭示更本质的关系。第三,我们需要进行语言建模,以提取存在于文本中的人类知识和概念。
接着,叶启威介绍了基于三维分子结构预训练和基于结构的预训练模型等生物分子表示方法。
叶启威团队在预训练范式方面进行了创新,改变了传统的方法。在小分子预训练的基础上,他们尝试了一种新的方法,将任意的局部结构首先建模成一个特定的四面体,并在这个新的范式中给这个四面体添加一些噪音。然后引入了去噪训练目标,通过对这类局部结构不断进行扰动和重构,可以得到更加鲁棒的生物分子的三维空间模型。此外,他们还尽量减少了先验假设的引入。例如,在图神经网络中,Message Passing不可避免地会压缩很多信息,因此他们使用了Transformer进行建模,并发现它在如分子生化性质预测等下游任务上表现更好。
叶启威团队还将上述方法扩展到蛋白质表示。由于蛋白质是大分子,因此他们使用蛋白质的碳骨架来表示蛋白质。
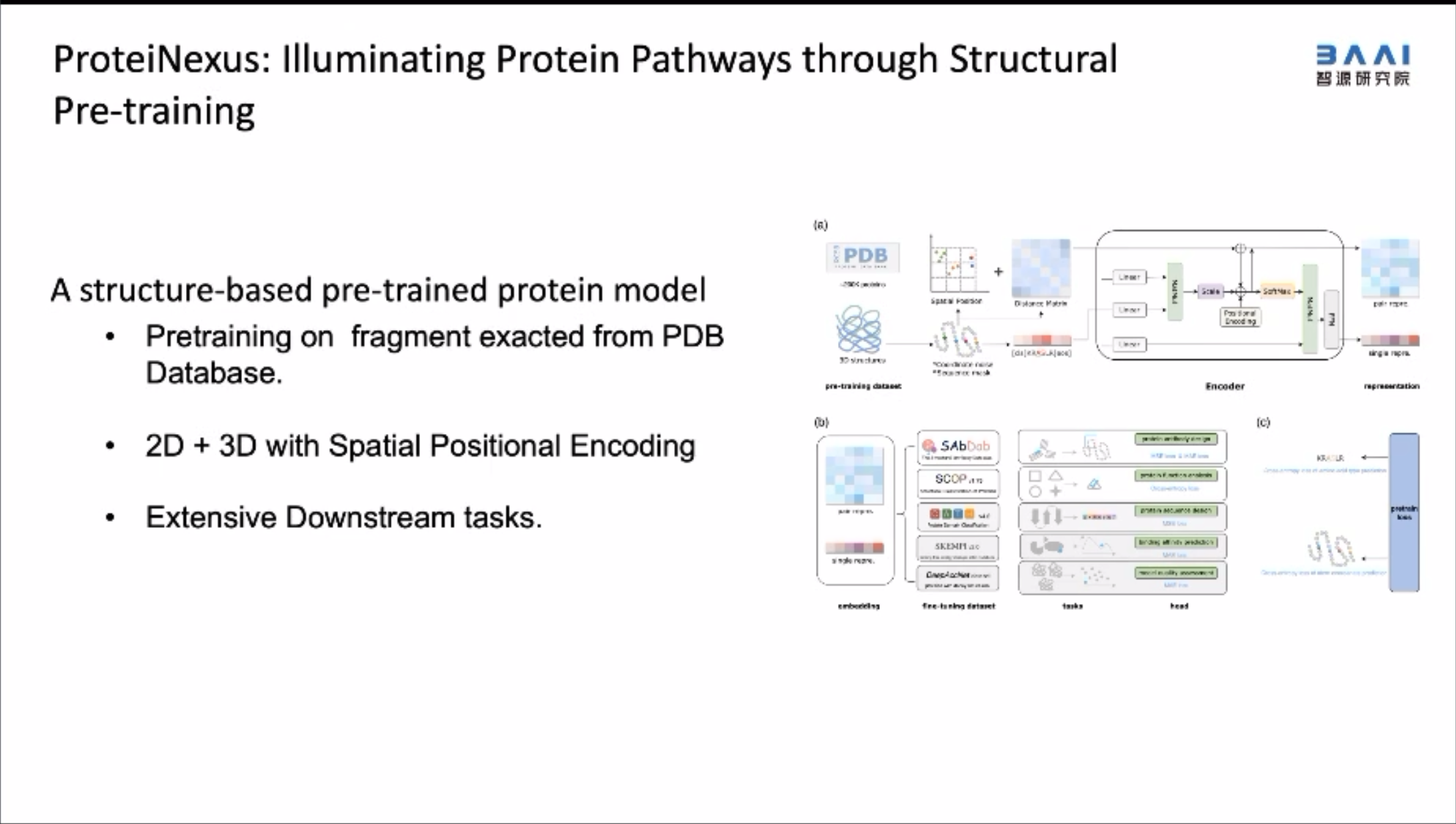
在建模过程中采用了SPE的方法,同时捕捉序列信息和结构信息。在构建不同蛋白原子之间的相互关系时,引入了欧几里得坐标系,并构建了一个适配于3D信息的位置的编码器,以有效地捕捉相互作用关系。通过在各种下游任务上进行finetuning,该模型在各种任务中都非常有效。
此外,叶启威团队还基于大型语言模型构建了与生物信息领域相关的对齐工作,让大型模型赋能生物分子领域,并在下游生化性质预测任务上取得了提升。
团队开源了OPI1.0,该工作通过对Swiss-Prot数据库和相关文献的抽取,获取了大量的关系数据,并将其转化为相应的指令。此外,他们还在LLAMA模型上进行了一些finetuning,并发现它对于定义的七个任务都表现出色。
此外团队还有计划对OPI进行进一步的扩展,涉及更为广泛的分子以及模态。
叶启威团队的第一阶段项目Opencomplex在蛋白质复合物上的表现超过了Alphafold2 Multimer,并在过去的一年中获得了CAMEO连续预测竞赛的年度冠军。此外,在CASP15的竞赛中,OpenComplex也取得了RNA结构预测的自动化赛道第一。叶启威指出,与传统的基于力场的方案相比,目前基于数据驱动的方法仍然存在优化的空间。
长远来看,叶启威总结道,我们的目的是基于底层人工智能技术和数据,构建一个强大的数字孪生模型,能够逐步从原子、分子、细胞层面来模拟生物分子之间的相互作用关系,为生命科学的发展提供全新的范式。